CUSTOMER
客户中心
助力生命健康领域从基础研究到产业化的加速转化
Abstract
The identification of patient-derived, tumor-reactive T cell receptors (TCRs) as a basis for personalized transgenic T cell therapies remains a time- and cost-intensive endeavor. Current approaches to identify tumor-reactive TCRs analyze tumor mutations to predict T cell activating (neo)antigens and use these to either enrich tumor infiltrating lymphocyte (TIL) cultures or validate individual TCRs for transgenic autologous therapies. Here we combined high-throughput TCR cloning and reactivity validation to train predicTCR, a machine learning classifier that identifies individual tumor-reactive TILs in an antigen-agnostic manner based on single-TIL RNA sequencing. PredicTCR identifies tumor-reactive TCRs in TILs from diverse cancers better than previous gene set enrichment-based approaches, increasing specificity and sensitivity (geometric mean) from 0.38 to 0.74. By predicting tumor-reactive TCRs in a matter of days, TCR clonotypes can be prioritized to accelerate the manufacture of personalized T cell therapies.
摘要
将患者来源的肿瘤反应性T细胞受体(TCR)识别作为个性化转基因T细胞治疗的基础,目前仍是一项耗时且成本高昂的工作。目前识别肿瘤反应性TCR的方法是分析肿瘤突变以预测激活T细胞的(新)抗原,并利用这些抗原富集肿瘤浸润性淋巴细胞(TIL)培养物,或者验证个体TCR用于自体转基因治疗。在此,我们结合高通量TCR克隆和反应性验证,训练了predicTCR,这是一种机器学习分类器,能够在不依赖抗原的情况下,基于单TIL RNA测序识别个体肿瘤反应性TIL。与以往基于基因集富集的方法相比,predicTCR能够更好地识别来自多种癌症的TIL中的肿瘤反应性TCR,将特异性和敏感性(几何平均值)从0.38提高到0.74。通过在几天内预测肿瘤反应性TCR,可以优先选择TCR克隆型,从而加速个性化T细胞治疗的生产。
内容总结
研究背景
从患者体内识别出肿瘤反应性T细胞受体(TCR)是开发个性化转基因T细胞疗法的基础,但这一过程目前既耗时又成本高昂。
传统的识别方法依赖于分析肿瘤突变以预测T细胞激活的(新)抗原,并利用这些抗原富集肿瘤浸润性淋巴细胞(TIL)或验证个体TCR。
研究方法
研究者结合高通量TCR克隆和反应性验证,训练了 predicTCR,这是一个能够在不依赖抗原的情况下,基于单TIL RNA测序识别个体肿瘤反应性TIL的机器学习分类器。
使用单细胞RNA测序(scRNA-seq)和VDJ测序(scVDJ-seq)技术,从肿瘤样本中获取TIL,并建立相应的肿瘤细胞系(BT21)用于实验验证。
实验设计
选择了一个62岁男性的脑转移瘤样本(BT21),该患者之前被诊断为黑色素瘤。通过全外显子测序确认BT21细胞系是原发肿瘤的良好模型。
从TIL中克隆了50个最频繁的TCR克隆型,并通过流式细胞术检测这些TCR对BT21细胞系的反应性,发现其中34个TCR具有肿瘤反应性。
使用这些实验数据训练 predicTCR50 分类器,并通过贝叶斯优化调整超参数,最终通过Fisher-Jenk自然断裂法确定TCR克隆型的肿瘤反应性阈值。
关键结果
predicTCR50 在预测BT21样本中的肿瘤反应性TCR方面表现优异,AUC值达到0.92,准确率达到0.91。
与基于基因集富集的签名方法相比,predicTCR50 在预测肿瘤反应性TCR方面表现更好,例如NeoTCR8(AUC 0.87,准确率0.50)、Hanada(AUC 0.77,准确率0.72)等。
在COVID-19患者PBMC样本中,predicTCR 没有错误地将任何T细胞分类为肿瘤反应性,显示出低假阳性率。
predicTCR 在多种肿瘤类型(包括胰腺癌、结直肠癌转移、非小细胞肺癌和胃肠道癌)的外部数据集上进行了验证,表现优于现有的签名方法,平均G-mean从0.38提高到0.74。
结论
predicTCR 是一个能够快速、准确地从多种癌症类型的TIL中识别出肿瘤反应性TCR的自动化分类器。
通过机器学习模型和确定性阈值的结合,predicTCR 能够在几天内预测肿瘤反应性TCR,从而加速个性化T细胞疗法的制造。
这种方法不仅提高了预测的准确性,还降低了成本,有望推动个性化T细胞疗法的临床应用。
图文摘要
图1 | BT21细胞系准确模拟切除的转移病灶,允许进行高信心的实验性TCR肿瘤反应性测试。
a. 实验和计算流程概述,支持predicTCR分类器:TILs被分类并进行scRNA+VDJ测序,同时邻近切除的肿瘤材料用于建立BT21肿瘤细胞系。TCR反应数据随后与scRNA+VDJ测序数据整合,以训练predicTCR分类器,随后在来自不同肿瘤类型的外部生成的TIL数据集上进行测试。
b. 从脑转移样本中恢复的TILs的scRNA-seq数据的无监督聚类(UMAP图),关键T细胞亚型被标注。
c. 将前20个TIL TCR克隆型的百分比频率及其分布投影到UMAP图上,显示相同克隆型的细胞可以占据多种表型状态。
d. 转染了50个最常见TIL衍生TCR克隆型之一的T细胞与BT21细胞共培养;通过流式细胞仪测量的CD107a水平(通过流式细胞仪量化,门控在表达转基因TCR的mTCRβ+细胞上)显示给定的TCR克隆型是否识别BT21细胞系。有关TCR反应阈值的详细信息,请参见方法。
e. 在TIL群体中,BT21反应性TCR克隆型比非反应性克隆型更频繁。
f. 将BT21反应性测试结果投影到UMAP图上(b)。

图2 | PredicTCR50分类器训练策略。
使用健康捐赠者的scRNA数据,以及来自样本BT21的50个最常见TCR TIL克隆型的scRNA+VDJ和实验性肿瘤反应数据,使用XGBoost训练中间模型。由于scRNA数据的稀疏性质,我们首先执行贝叶斯优化以使用分层k折交叉验证调整超参数。随后我们使用可解释的AI SHAP识别此中间模型中的关键特征(即基因),然后仅使用这些特征训练一个更简单的模型,以防止对训练数据过拟合。然后像以前一样对更简单的模型进行优化,并应用于剩余的BT21 TIL数据。分类器计算的每个细胞的反应概率被平均化,以计算每个TCR克隆型的Fisher-Jenk自然断点,以确定将TCR称为肿瘤反应的适当最低阈值。
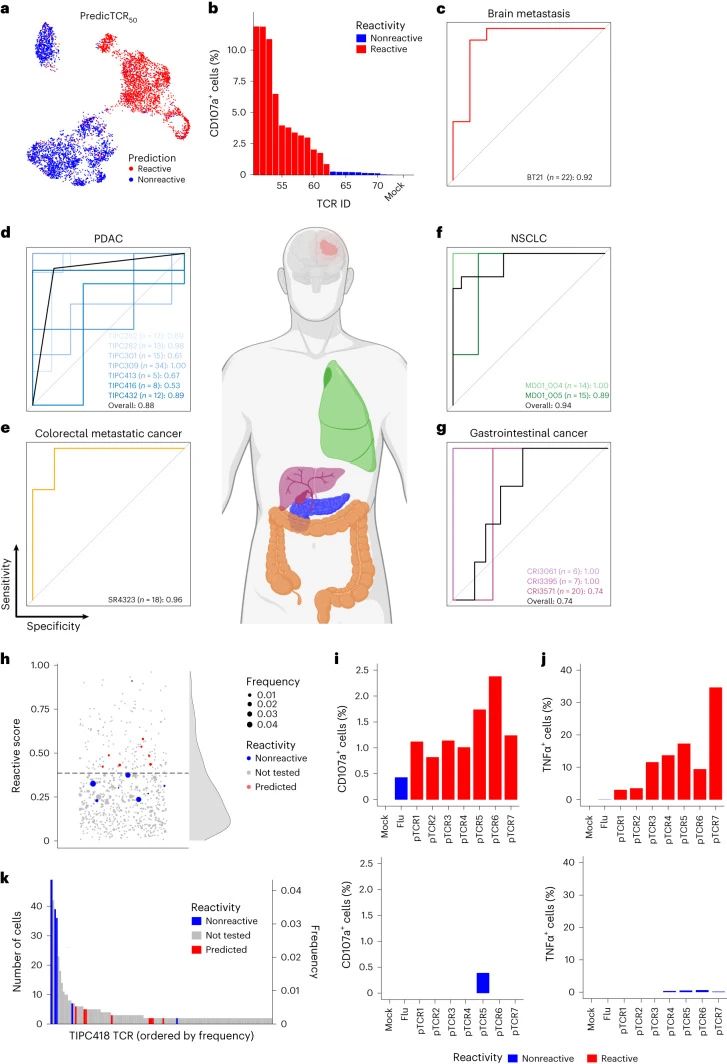
图3 | PredicTCR准确预测不同肿瘤类型的肿瘤反应性TCR。
a. 在图1中叠加PredicTCR50每个细胞的肿瘤反应性预测的UMAP图。
b. 另外测试了22个TCR克隆型(29个不同的TCR α/β链对)对BT21细胞系的反应性。
c. PredicTCR50在预测BT21患者肿瘤反应性TCR方面的性能。
d-g. PredicTCR在预测已发布的具有TCR反应性数据的scRNA+VDJ数据集中TCR肿瘤反应性方面的性能:Meng等人的7个PDAC样本(d),Caushi等人的一个结肠转移23(e),两个NSCLC19(f)和三个胃肠道癌症21(g)。每个样本的TCR克隆型数量和AUC值列出。所有可用TCRs按癌症类型评估的整体性能。
h. PredicTCR对Meng等人的PDAC样本TIPC418的再活动预测,该样本测试了8个TCRs,发现没有对TIPC418衍生的肿瘤细胞系反应(蓝色点,点大小按TIL TCR克隆型数量缩放)。PredicTCR分析预测这8个TCR中有7个是非反应性的(反应性分数低于Fisher-Jenk自然断点,图中的虚线)。预测为肿瘤反应性的7个额外TCR克隆型(红色点)被克隆以进行前瞻性验证。
i,j. 与TIPC418细胞共培养的表达预测的TIPC418反应性TCR的T细胞的流式细胞仪分析,通过CD107a(i)和TNFα(j)确认所有7个TCR都是反应性的。
k. 恢复的TILs的相对频率和绝对数量,针对在h-j中测试的TCR克隆型。
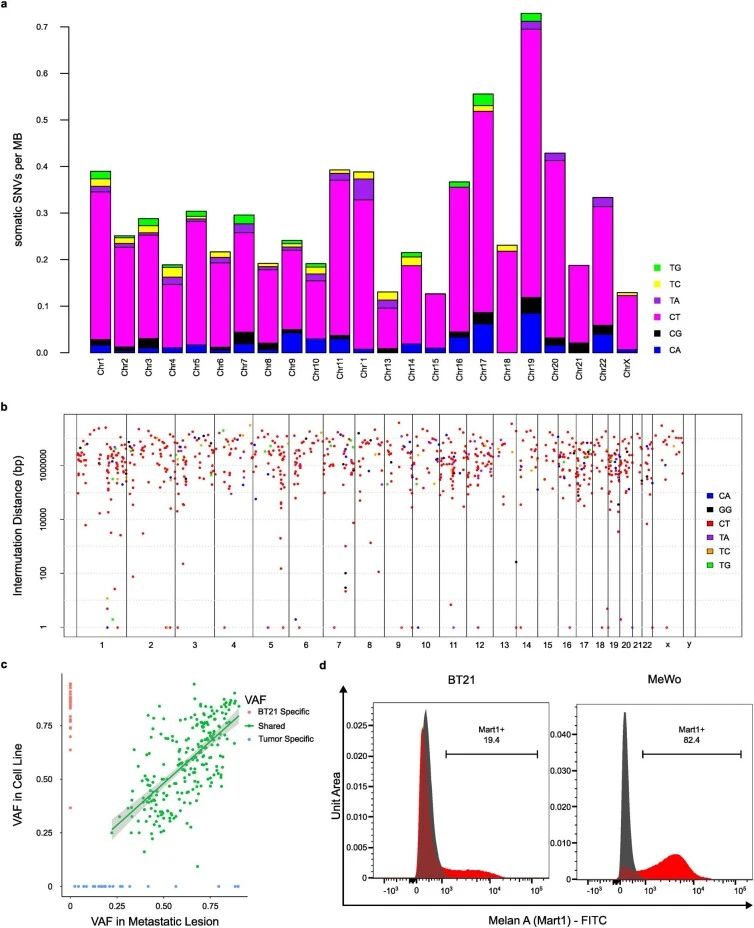
扩展数据图1 | BT21展示出黑色素瘤特征的突变模式。
a. 转移性黑色素瘤样本BT21显示出黑色素瘤典型的UV介导C>T转换突变的主导地位。
b. 突变在染色体上的可视化及其密度,显示突变遍布整个基因组,再次显示了原发性黑色素瘤的典型特征。
c. 变异等位基因频率(VAF)图显示BT21转移性肿瘤样本和BT21细胞系中发现的SNVS重叠;细胞系的额外突变可能是肿瘤异质性的结果或在适应细胞培养条件的过程中产生。完整的SNVSN数据以*.vcf格式包含在源数据1中。误差带代表残差的标准误差。
d. 流式细胞仪分析确认BT21细胞系表达肿瘤相关抗原MART1,如在90%的原发性皮肤黑色素瘤中发现的那样。常用的MeWo黑色素瘤细胞系显示为比较。
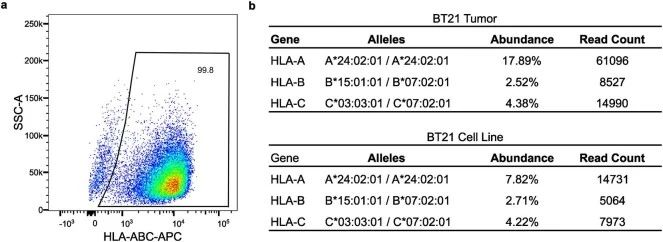
扩展数据图2 | BT21细胞系适合免疫测试。
a. BT21细胞系在用300 IU/mL IFNγ刺激两天后表达高水平的MHC I。
b. 使用arcasHLA进行的体外HLA分型*确认BT21肿瘤细胞系源自原发性肿瘤,并且在没有刺激的情况下表达HLA-A、-B和-C基因。‘丰度’指标表示RNA测序读数映射到HLA等位点的百分比,并给出了HLA位点之间表达的相对度量。‘读数’表示映射到等位点的RNA测序读数的绝对数量。
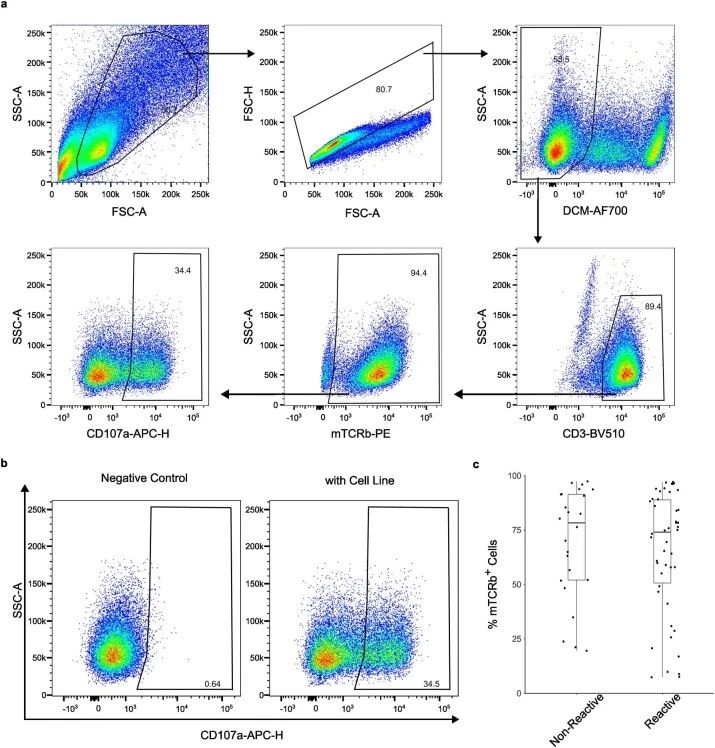
扩展数据图3 | 使用细胞系BT21的TIL衍生TCR测试数据。
a. TCR测试的门控策略。它被门控为淋巴细胞,单细胞,随后是活细胞,然后是CD3+ T细胞,mTCRb+转基因T细胞然后是CD107a用于反应性。
b. 展示流式细胞仪数据,显示最反应性BT21 TCR与BT21细胞系共培养时CD107a的量化。共培养后,34.5%的细胞对CD107a呈阳性。
c. 肿瘤反应性和非反应性TCR克隆型的膜表达无显著差异。中心线代表中位数,下和上须分别对应第一和第三四分位数,上须延伸到最大值和最小值(n = 26非反应性TCR,n = 46反应性TCR)。
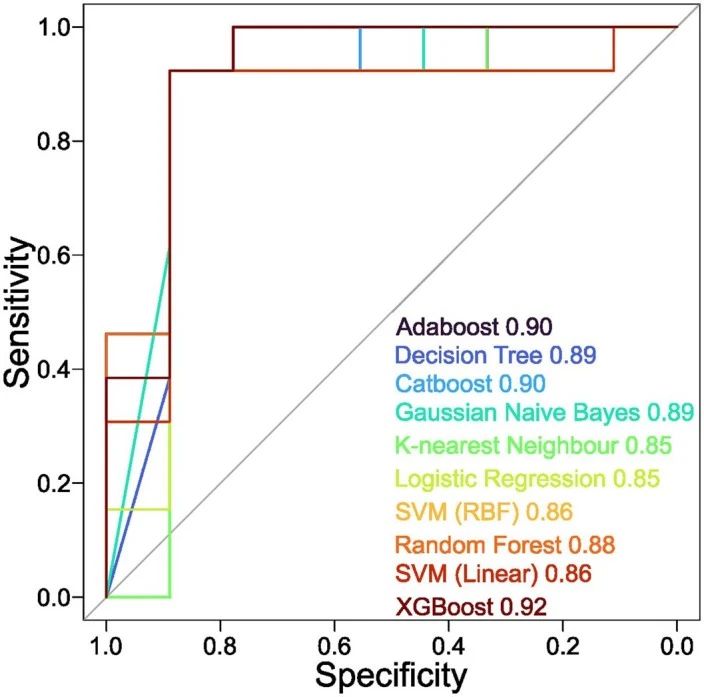
扩展数据图4 | 评估不同机器学习框架对TCR肿瘤反应性预测的性能。
所有模型均使用scikit-learn包实现,XGBoost来自xgboost包除外。AUC代表在没有进一步超参数调整的情况下模型性能。模型使用来自50个BT21 TCRs的数据进行训练。
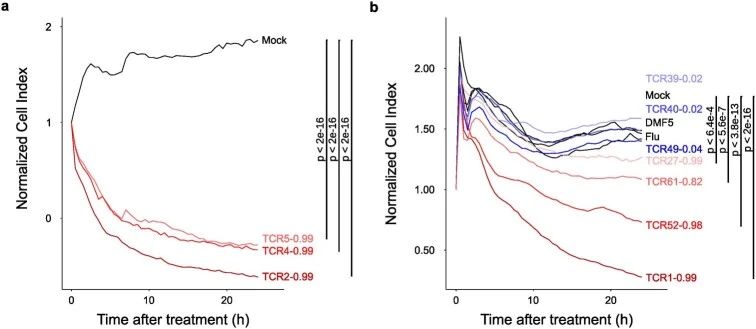
扩展数据图5 | 选择的BT21 TIL衍生TCRs可以在共培养试验中杀死BT21细胞系。
a. xCELLigence试验显示,与BT21细胞共培养时表达高水平CD107a的健康供体PBMCs转染有CD8+ TCR可以在共培养试验中杀死BT21细胞(图1d)。
b. 额外的共培养实验测试BT21衍生TCRs的杀伤能力,包括predicTCR50指定为肿瘤反应性的TCR。试验使用与a中相同的健康供体的PBMCs进行。线条是重复的平均值。统计分析作为单因素ANOVA在终点标准化细胞指数上进行,与模拟进行事后Dunnett测试。每个TCR克隆型的predicTCR50分数被计算并添加到图中。
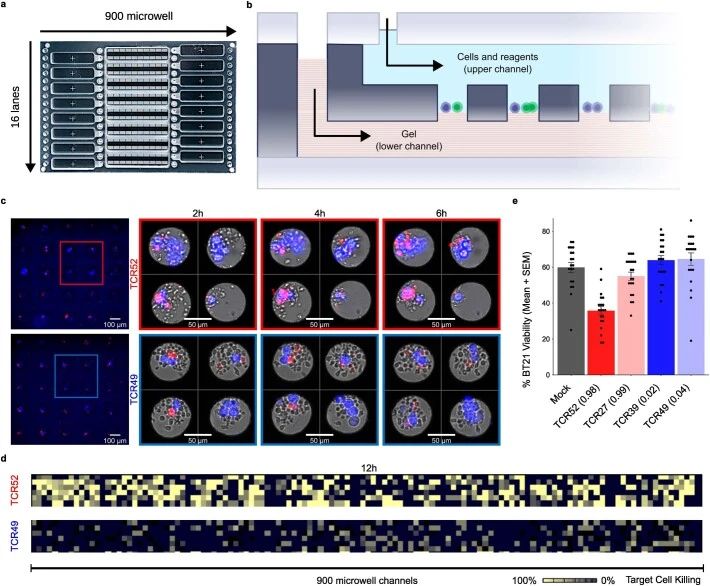
扩展数据图6 | 通过predicTCR识别的TCR的单细胞杀伤能力分析。
a. CC-Array微流体芯片的显微照片,由VivaCyteTM平台测量。
b. CC-Array设备的每一道包括一个加载水凝胶的较低微流体通道,一个上部微流体通道,用于输送细胞和试剂,以及一个对上下通道都开放的微孔阵列。一旦细胞加载到上部通道,它们就在凝胶界面处沉淀;随机加载导致每个微流体通道中效应细胞与肿瘤细胞比例的分布。
c. 显示共培养的PBMCs表达转基因TCR和BT21靶细胞的微孔子集的荧光图像,在三个时间点(每个时间点显示从900个复制微孔中抽取的4个微孔)。CM染色细胞(蓝色)上的PI信号(红色)随时间增加,反映了BT21靶细胞被转基因效应T细胞杀死。
d. 每个孔由VivaCyte软件量化,以产生每个TCR杀伤活性的概览。
e. 四个不同TCRs的结果量化重现了xCELLigence数据(扩展数据图5b),其中TCR52在12小时共培养后比TCR 27更有效地进行靶细胞杀伤。括号中的每个TCR显示predicTCR反应性分数。数据以平均值±SEM表示,对于单个实验运行,每个平均值代表20个视场(FOV)的平均值,每个FOV由平均45个微孔组成。
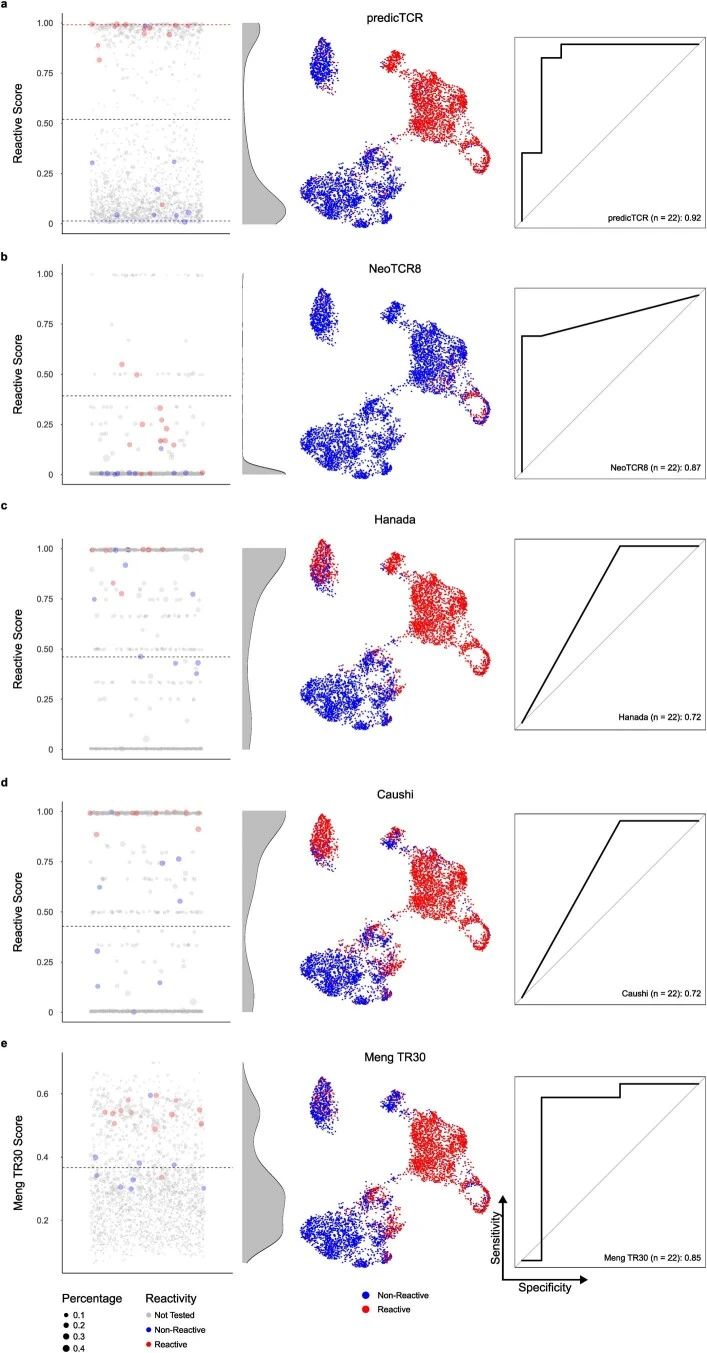
扩展数据图7 | 评估基于基因签名的TCR肿瘤反应性预测在BT21衍生TCRs上的整体性能。
每行的左侧面板显示了来自BT21的TIL衍生TCR克隆型的肿瘤反应性预测分数的平均分布,右侧有灰色阴影密度图。彩色点显示BT21验证集中的TCR克隆型(n = 22)。TCRs根据实验验证的反应性着色,每个点的大小反映该TCR克隆型在TILs中的频率。灰色虚线表示样本特定的自然断点,用于将肿瘤反应性与非反应性克隆型分开,而红色和蓝色虚线分别代表面板(a)中肿瘤反应性分数的95百分位和5百分位。中央面板将每个分类器的每细胞预测投影到图1b中的UMAP图上。右侧面板显示每个分类器的ROC曲线和AUC值,详细信息见表1。
a. predicTCR50的性能,显示出清晰的双峰分布分数,正确预测222个验证集TCRs的反应性。注意,假阳性预测(蓝色点靠近图顶部)位于95%阈值线以下。
b. Lowery等人的NeoTCR8的性能显示对222个TCR测试集的肿瘤反应性预测不佳。
c. Hanada等人开发的签名表现高假阳性检测率;注意预测顶部左侧簇(调节性T细胞,见图1b)中的肿瘤反应性T细胞。
d. Caushi等人开发的签名在BT21 TILs上也显示出类似的高假阳性率。e:Meng等人的签名TR30的性能比predicTCR50有更高的假阳性率。
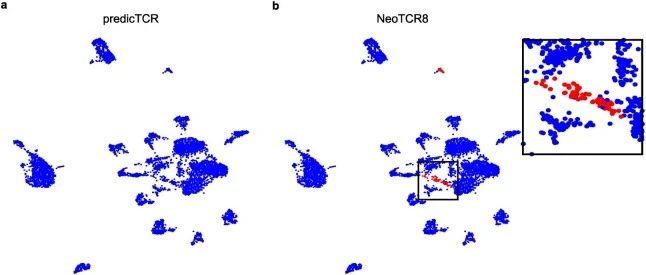
扩展数据图8 | 显示predicTCR和NeoTCR8在严重症状COVID-19感染患者(PP4)的PBMCs上的性能。
a. predicTCR在PP4患者PBMCs上的性能,没有细胞预测为肿瘤反应性。
b. 同一患者的NeoTCR8性能;插图面板显示包含T细胞(n = 130,占总细胞的2%)的簇的放大视图,被预测为肿瘤反应性。PBMC数据来自Yoshida等人。
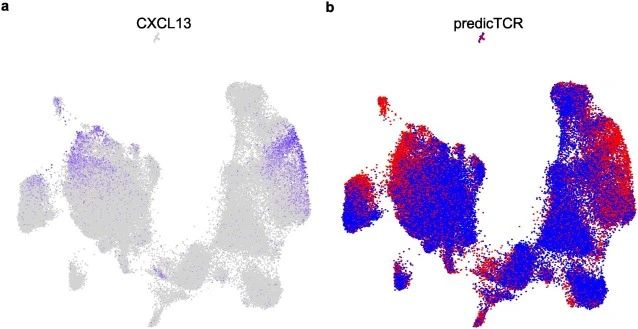
扩展数据图9 | UMAP显示CXCL13表达并不总是决定TILs的一个明确簇,但与predicTCR预测相似。
a. 在MD01-005患者中表达CXCL13的T细胞在降维后并未形成一个独立的簇;这加强了从基于簇的基因集富集分析中获得准确结果的困难。
b. predicTCR肿瘤反应性预测(这也部分依赖于CXCL13)不局限于单一簇,并且包括(但不限于)表达CXCL13的细胞。
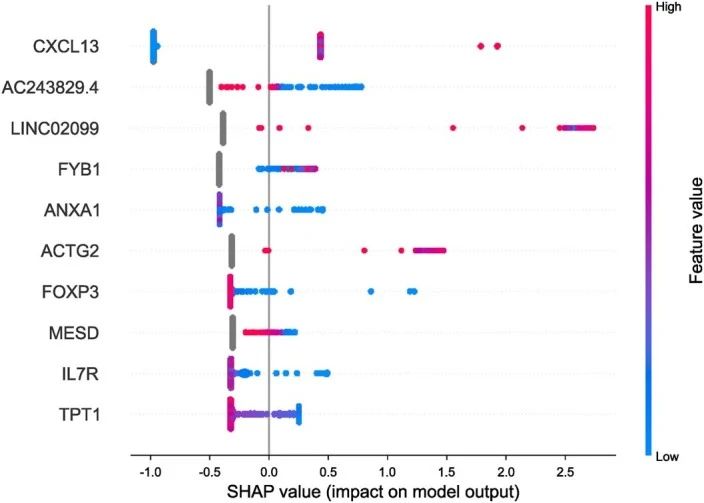
扩展数据图10 | 可解释的AI SHAP对predicTCR的分析。
对predicTCR分类器执行了SHAP,以识别对分类器有贡献的前10个特征(基因)。前10个基因被展示为蜂群图,每个点代表一个单一细胞。颜色表示特定基因的表达值,x轴表示SHAP值,即特定特征对模型输出的影响。正SHAP值有助于反应性预测,反之亦然。
参考文献:
Tan CL, Lindner K, Boschert T, Meng Z, Rodriguez Ehrenfried A, De Roia A, Haltenhof G, Faenza A, Imperatore F, Bunse L, Lindner JM, Harbottle RP, Ratliff M, Offringa R, Poschke I, Platten M, Green EW. Prediction of tumor-reactive T cell receptors from scRNA-seq data for personalized T cell therapy. Nat Biotechnol. 2025 Jan;43(1):134-142. doi: 10.1038/s41587-024-02161-y. Epub 2024 Mar 7. PMID: 38454173; PMCID: PMC11738991.