CUSTOMER
客户中心
助力生命健康领域从基础研究到产业化的加速转化
皮下成瘤模型算是肿瘤研究里最常见的一类,入门简单,但真正要把数据整理到“可以投稿”的水准,很多人都会卡壳:曲线画出来了,终点肿瘤也称了,HE也染了几张,结果一到写文章,图版拼不顺,机制说不完整,整体显得又散又虚。
问题往往出在一开始,没有按“未来的图版结构”设计实验和取材。下面以皮下成瘤为例,从建模开始,把一整套能直接复用的实操流程分享给大家。
从建模开始,就按“图1”的思路采数据
皮下成瘤的第一层任务,是把体内主效应站稳,这就是文章里的“图1”。
具体可以这样做:
一个是“早期时间点”,例如第7–10天,用来看机制启动阶段;
一个是“终点时间点”,用来看最终效应。
这样最后至少会拿到三类体内数据:
完整的生长曲线、终点肿瘤重量、早期和终点两个节点的组织。到这里,图1就有了基础:生长曲线+终点重量+瘤体代表照片,再加一条体重曲线做安全性背景。
肿瘤的取材保存至关重要
很多课题组最大的问题,是取材时习惯“一刀下去整块丢福尔马林”,后面要WB、qPCR、ELISA才发现组织不够用,或者只能从别的动物补,数据一致性就开始打折扣。
更稳妥的做法是:
每只肿瘤,都当成一块要被拆分利用的综合资源,而不是一块“病理标本”。
实际操作可以这样安排:
第一半直接放到4%多聚甲醛,后面做HE、Ki67、TUNEL、CD31这类病理和免疫组化;第二半再分成几小块,用铝箔包好,快速丢到液氮罐里冻住,之后分别用于WB、qPCR、ELISA或其他检测。
这样一只瘤就能支持整整三层证据:组织结构、蛋白水平、mRNA或细胞因子。
体内主效应怎么做成“站得住脚的图1”
图1的目标很简单:让任何人第一眼就看懂,你的处理对肿瘤生长有没有实打实的影响。
通常包括以下内容:
做到这个程度,体内主效应基本合格。真正拉开差距的是后面几层数据能不能有机接上这一层。
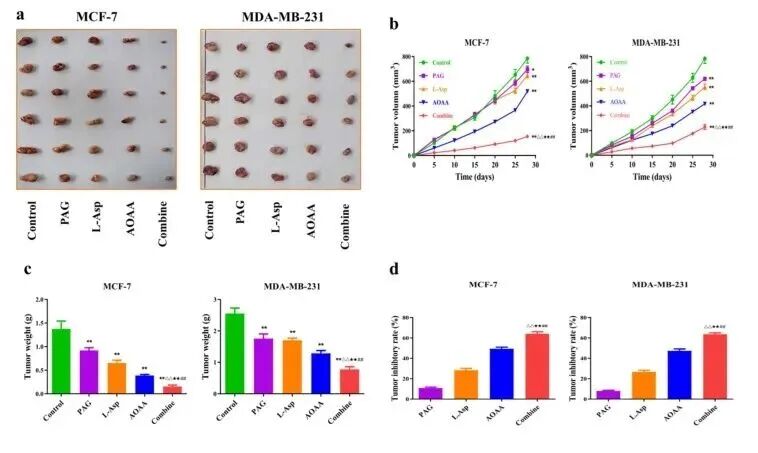
(doi: 10.3390/molecules27134049,Figure 6 )
病理和免疫组化:组织层要讲清瘤体差异
瘤体变大或变小只是现象,对应的原因必须在组织层解释清楚。
常见的组合设计如下:
HE:看整体结构、细胞密度、坏死区。
Ki67:反映增殖状态,阳性细胞比例和肿瘤生长速度往往对应。
TUNEL或cleaved-caspase-3:反映凋亡。
CD31:看血管密度,为“供血不足导致生长受限”这类解释提供依据。
如果做免疫相关:
可以用CD8、F4/80、CD206等标记来说明免疫浸润情况。
关键点在于染出来的结果能不能和体积变化对得上。
例如:肿瘤长得慢的那组,Ki67减少、TUNEL增多、CD31下降,这三者一起出现,比单独一张HE图有说服力得多。
定量时最好用ImageJ之类的软件做阳性面积或阳性细胞计数,最后以柱状图形式呈现,和体内体积、重量放在一前一后。
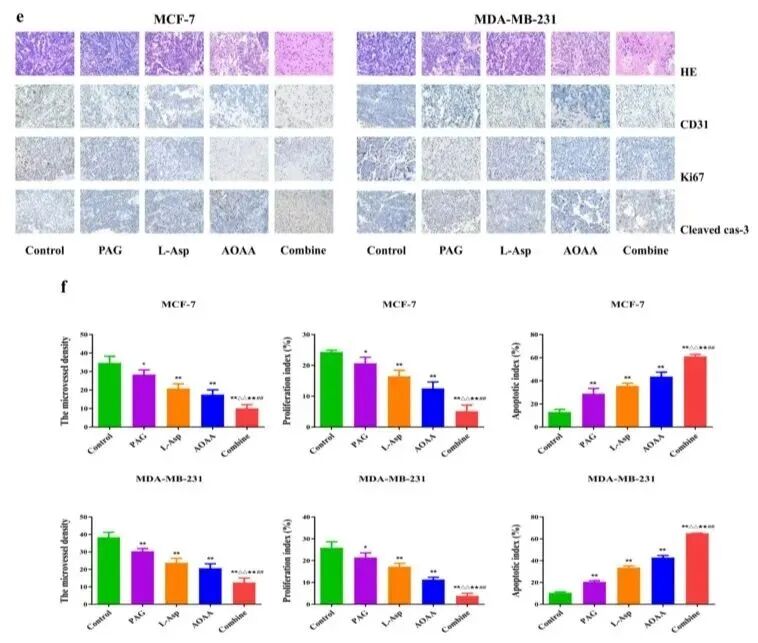
(doi: 10.3390/molecules27134049,Figure 6)
机制层:从组织结果往回倒推要测什么
到了机制层,很多文章会开始“迷路”,测了一堆蛋白,张张略有差异,但没有一条足够清晰的通路。
更有效的思路是先看组织层告诉了你什么,再根据这些变化选通路。
如在组织层已经观察到到增殖下降、凋亡上升:
那机制上可以重点围绕AKT/ERK通路、细胞周期蛋白、Bcl-2家族、caspase通路展开。
如果文章走铁死亡方向:
可以把GPX4、SLC7A11、ACSL4、FTH1等放进WB;
再配合MDA、GSH、铁含量测定,组织层的脂质过氧化染色。
做代谢类课题时:
通常会选HK2、LDHA、PKM2、CPT1A、PGC1α等关键酶或代谢通路节点。
机制层的目标很明确:
把体内、组织层的变化压缩成一条两三步的分子通路。每条通路选3–5个真正关键的蛋白或mRNA即可。

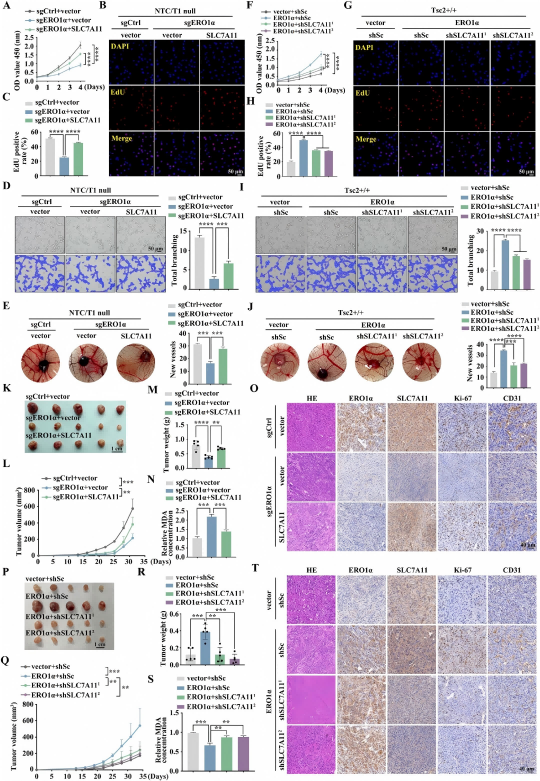
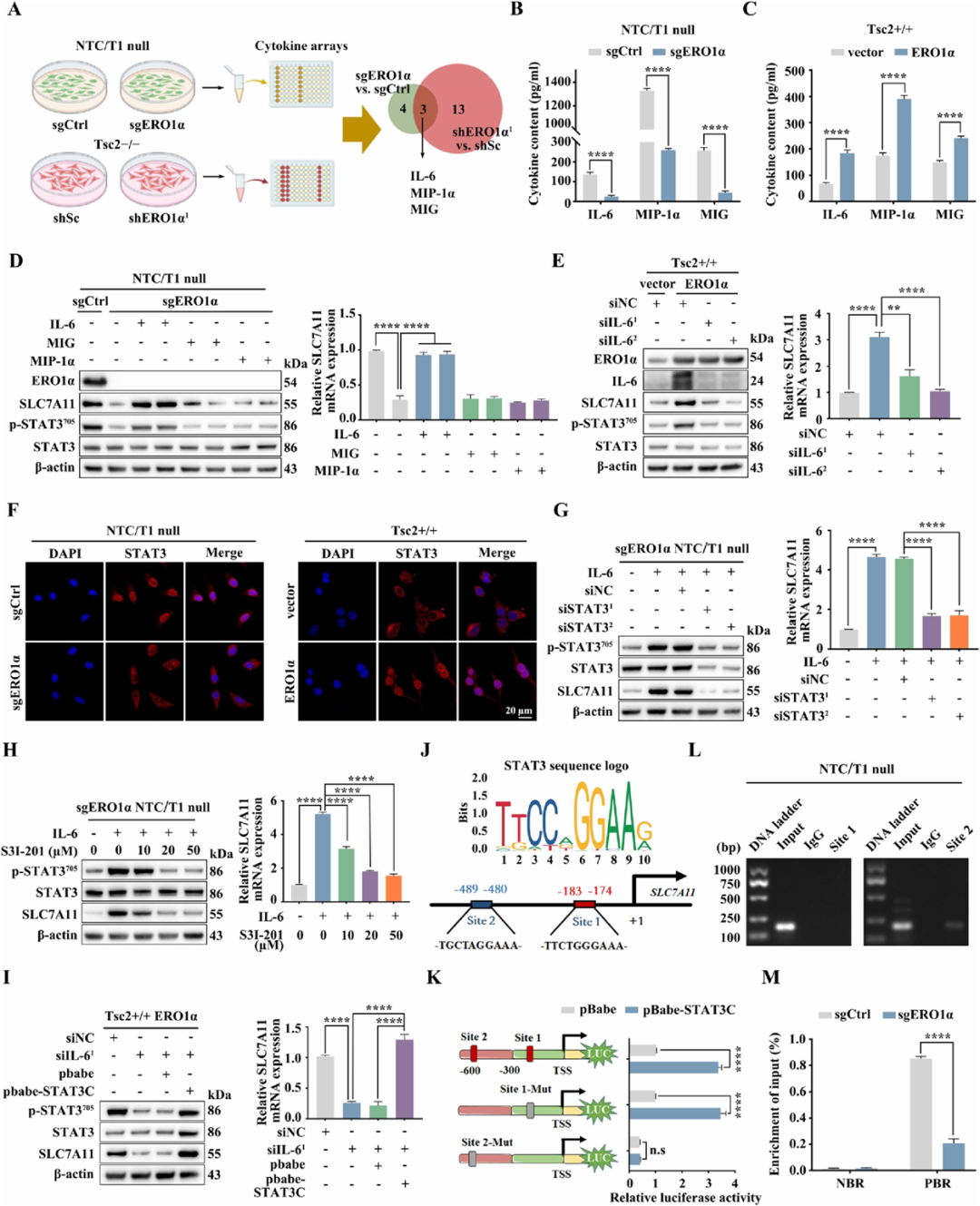
(DOI:10.1186/s13046-024-03039-2,Figure 4,5,6)
体外验证:只做关键几刀,把因果补全
体内机制的“相关性”通常可以这样安排:
只要体外结果和体内机制方向一致,并且有rescue,把“某通路确实决定这个表型”这件事说明白,文章的逻辑链基本就闭合了。
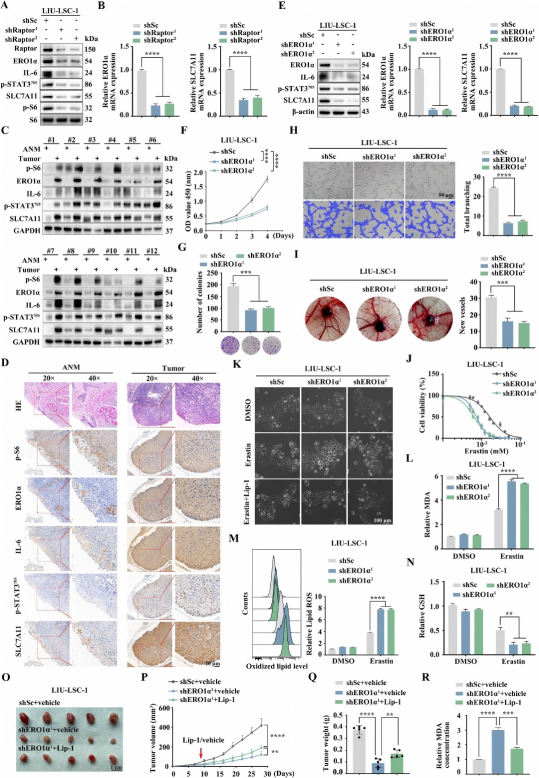
(DOI:10.1186/s13046-024-03039-2,Figure 7)
图版怎么排,才能一眼看出结构完整
很多人做完所有实验才开始想图版怎么拼,结果要么挤在一起,要么顺序混乱。更省事的方式,是直接用一套固定模板,后面做任何皮下成瘤课题,都往里面填内容即可。
整体可以按这样的顺序来:
图1做体内主效应:成瘤曲线、终点重量、代表照片、体重变化,交代清楚“有没有抑瘤”。
图2做组织层:HE、Ki67、TUNEL、CD31和可能的免疫浸润标记,说明瘤体变化来源。
图3做体内机制:肿瘤组织的WB、qPCR、ELISA,把通路趋势画出来,接上组织层。
图4做体外功能验证:细胞实验的增殖/凋亡/迁移结果,对应同一条通路。
如果课题复杂,再加一张图5放rescue或更深入的机制扩展。
只要按照这个框架搭,一般审稿人翻到图3基本就能明白你想说什么,读起来也不会觉得散。
可复用的皮下成瘤科研配图整理SOP
这套逻辑换一套细胞系、换一种药物、换一个通路依然成立。只要文章还在用皮下成瘤模型,那么这个SOP就能一直复用。
如果后面你想把某个具体方向(比如铁死亡、免疫微环境、代谢重编程)放进这套框架,也可以在机制部分做一次针对性替换,文章结构也不用推倒重来。