CUSTOMER
客户中心
助力生命健康领域从基础研究到产业化的加速转化
From: Nature Communications,IF=16.1

Abstract
Single-cell metabolomics reveals cell heterogeneity and elucidates intracellular molecular mechanisms. However, general concentration measurement of metabolites can only provide a static delineation of metabolomics, lacking the metabolic activity information of biological pathways. Herein, we develop a universal system for dynamic metabolomics by stable isotope tracing at the single-cell level. This system comprises a high-throughput single-cell data acquisition platform and an untargeted isotope tracing data processing platform, providing an integrated workflow for dynamic metabolomics of single cells. This system enables the global activity profiling and flow analysis of interlaced metabolic networks at the single-cell level and reveals heterogeneous metabolic activities among single cells. The significance of activity profiling is underscored by a 2-deoxyglucose inhibition model, demonstrating delicate metabolic alteration within single cells which cannot reflected by concentration analysis. Significantly, the system combined with a neural network model enables the metabolomic profiling of direct co-cultured tumor cells and macrophages. This reveals intricate cell-cell interaction mechanisms within the tumor microenvironment and firstly identifies versatile polarization subtypes of tumor-associated macrophages based on their metabolic signatures, which is in line with the renewed diversity atlas of macrophages from single-cell RNA-sequencing. The developed system facilitates a comprehensive understanding single-cell metabolomics from both static and dynamic perspectives.
摘要
单细胞代谢组学能够揭示细胞异质性并阐明细胞内分子机制。然而,传统的代谢物浓度测量仅能提供代谢组的静态快照,缺乏对生物通路代谢活性的动态信息。本研究开发了一种基于稳定同位素示踪的单细胞动态代谢组学通用系统,整合了高通量单细胞数据采集平台与非靶向同位素示踪数据处理平台,形成单细胞动态代谢组学的完整工作流程。该系统可在单细胞水平实现全局代谢活性描绘与复杂代谢网络的通量分析,并揭示单细胞间代谢活性的异质性。其重要性在2-脱氧葡萄糖抑制模型中得到凸显——该系统可检测出浓度分析无法反映的细微代谢变化。尤为关键的是,结合神经网络模型,该系统实现了对直接共培养肿瘤细胞与巨噬细胞的代谢组学分析,揭示了肿瘤微环境中复杂的细胞间互作机制,并首次基于代谢特征鉴定出肿瘤相关巨噬细胞的多功能极化亚型,与单细胞RNA测序重构的巨噬细胞多样性图谱高度吻合。本系统为从静态与动态双重维度全面理解单细胞代谢组学提供了有力工具。
内容总结
单细胞代谢异质性揭示
MDA-MB-231细胞模型:在5000个单细胞中识别出6个代谢亚群(A–F),表现出非同步的代谢响应和异质性代谢速率,传统群体分析无法捕捉。
代谢通路互联分析:揭示了UDP-葡萄糖/UDP-半乳糖在糖酵解、戊糖磷酸途径、核苷酸代谢间的动态通路切换,还原性TCA循环与氧化性TCA循环的细胞间差异。
2-DG药物扰动实验
代谢活性 vs 代谢物浓度:即使在药物处理后代谢物浓度未变,代谢通量已显著下降(如UDP-GlcNAc),证明动态代谢组学比静态浓度更敏感。
代偿机制:糖酵解抑制后,细胞通过谷氨酰胺回补TCA循环、增强丙酮酸羧化酶通路等方式维持能量代谢。
肿瘤-巨噬细胞共培养模型
细胞身份识别:构建神经网络模型,无需荧光标记即可从代谢特征中准确区分肿瘤细胞(MDA-MB-231)与巨噬细胞(THP-1来源),准确率>90%。
代谢重编程
肿瘤细胞:上调糖酵解、戊糖磷酸途径、核苷酸糖代谢;下调谷氨酰胺代谢与氨基酸合成。
巨噬细胞(TAM):表现出M2样极化特征,如氧化磷酸化、TCA循环、精氨酸合成通量增强;UDP-GlcNAc积累提示其可能调控M1/M2平衡。
TAM亚群精细分型
4个代谢亚型(基于代谢活性):
Cluster 1/2:氧化磷酸化与精氨酸合成活跃,对应促血管生成与EMT诱导功能。
Cluster 3/4:UDP-GlcNAc代谢活跃,参与ECM重塑与干扰素信号通路。
与scRNA-seq数据整合:验证这些代谢亚型与已报道的TAM基因亚群(如RTM_int、Mac_Angio、Mac_IFN等)具有功能一致性。
结论与意义
技术层面:首次实现单细胞水平的动态代谢通量分析,为研究细胞异质性、药物响应、微环境交互提供了新工具。
生物学层面:揭示肿瘤-巨噬细胞互作中的双向代谢重塑,并提出TAM极化不仅限于M1/M2二分法,而是连续、异质的代谢状态谱。
图文摘要
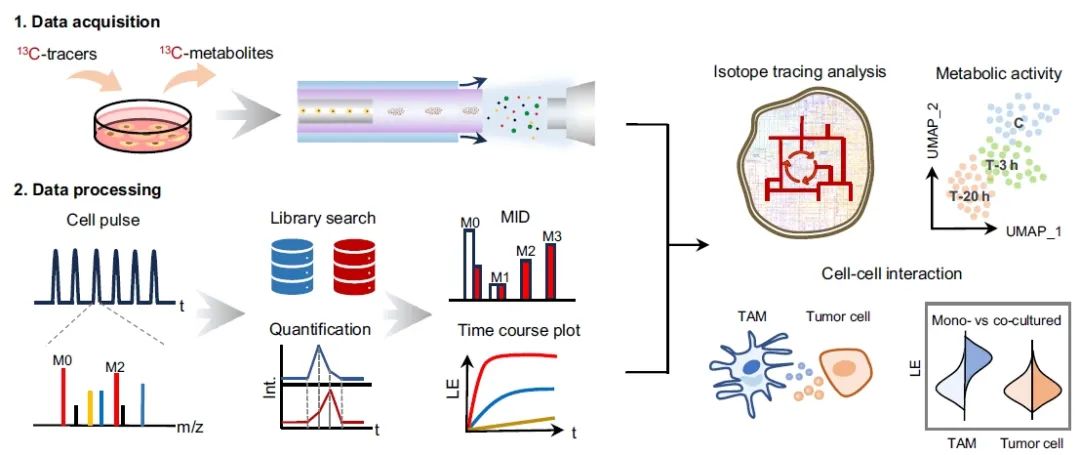
图1. 动态单细胞代谢组学系统流程示意图。
细胞用同位素示踪剂标记后,通过高通量数据采集平台进行分析。随后,基于 Python 的数据处理平台完成单细胞关键数据处理与同位素示踪分析。该系统可在单细胞水平解析代谢活性,并对交错代谢网络进行通量分析,进而应用于细胞-细胞相互作用研究。
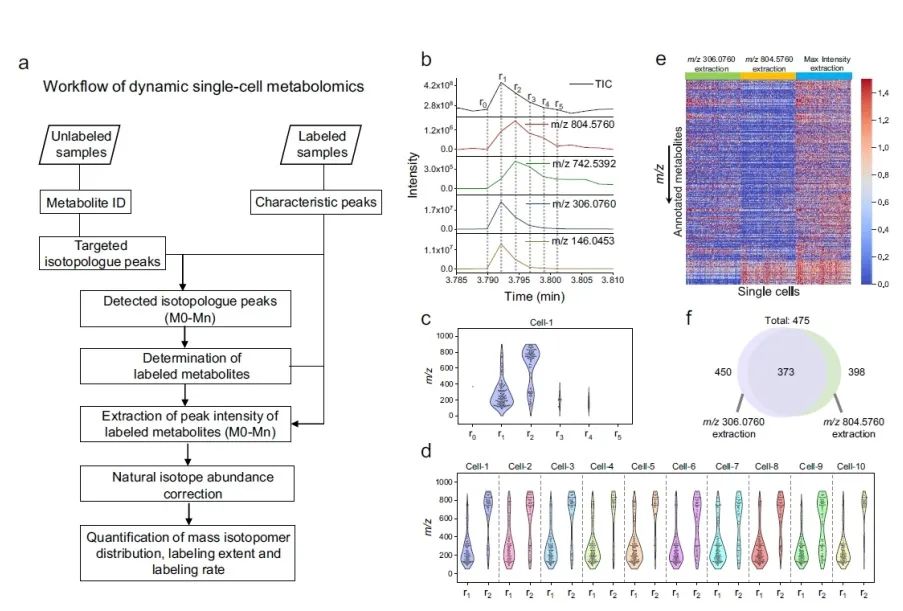
图2. 动态单细胞代谢组学数据处理平台。
a. 动态单细胞非靶向代谢组学数据处理流程。
b.单个细胞脉冲的总离子流图(TIC)与细胞标记物(m/z 306.0760、804.5760)的提取离子流图(EIC)。
c.一次细胞脉冲中所有代谢物最大强度对应的保留时间(RT)分布(r0–r5)。
d.10 个单细胞中不同 m/z 代谢物最大强度 r1/r2 比值分布。
e.用指定细胞标记或最大强度提取的单细胞代谢物相对强度热图。数据按每行 m/z 的均值进行归一化。
f.分别通过两个细胞标记提取的已注释代谢物的 Venn 图。所用细胞系为 MDA-MB-231,细胞数 n = 188。
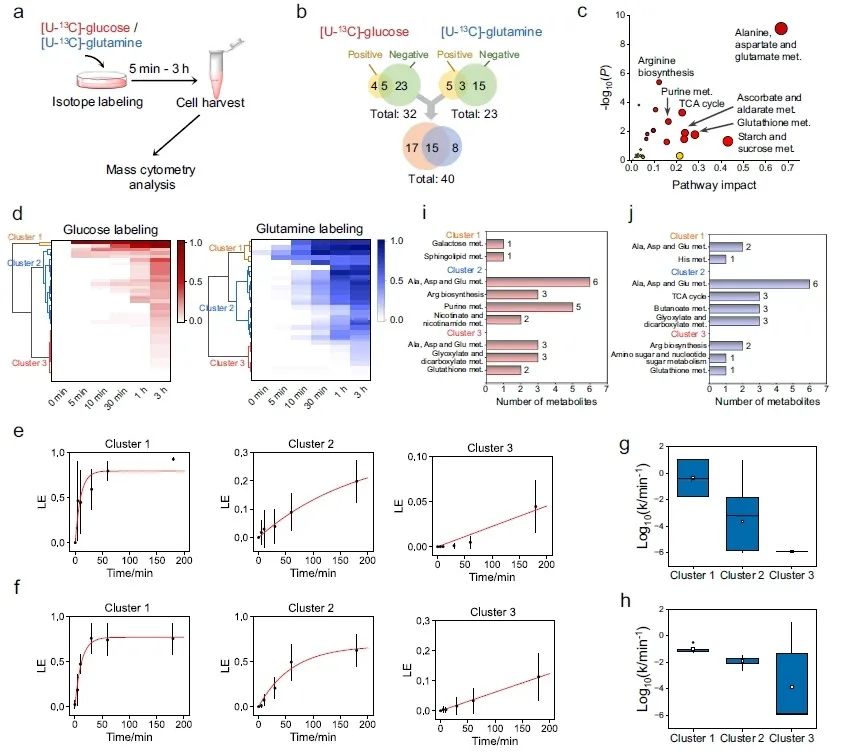
图3. 全球单细胞代谢活性谱分析。
a.细胞同位素标记及质谱流式分析示意图。
b.分别在 [U-¹³C]-葡萄糖与 [U-¹³C]-谷氨酰胺标记、正/负离子模式下检出的标记代谢物 Venn 图。
c.标记代谢物的 KEGG 富集分析。
d.不同时间点各标记代谢物平均标记程度(LE)的分层聚类分析。
e-f. 分别用 (e) [U-¹³C]-葡萄糖 和 (f) [U-¹³C]-谷氨酰胺 示踪的 3 个聚类中代谢物 LE 随时间变化曲线。数据以均值±SD(黑点)表示;红线为指数拟合曲线。
g-h. 分别用 (g) [U-¹³C]-葡萄糖 和 (h) [U-¹³C]-谷氨酰胺 示踪的 3 个聚类中各代谢物对数化标记速率 k 的箱线图。箱线中心为中位数,箱体上下缘为 25% 与 75% 分位数,须为 1.5×IQR 范围内的最大/最小值;白点为均值。
i-j. 分别用 (i) [U-¹³C]-葡萄糖 和 (j) [U-¹³C]-谷氨酰胺 示踪的 3 个聚类中代谢物的 KEGG 通路注释。每组 n = 100 个细胞。
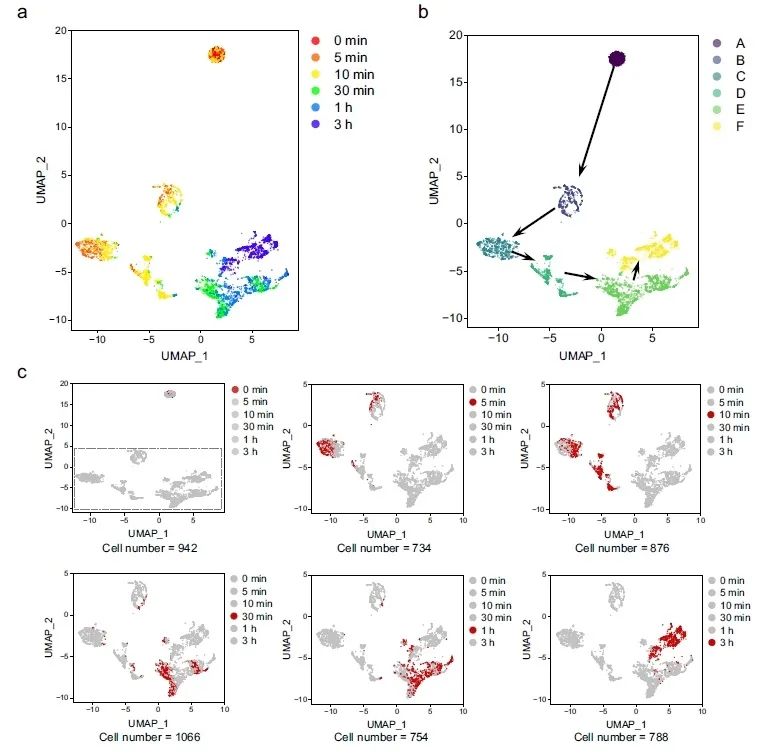
图4. 单个MDA-MB-231细胞的代谢活性细胞异质性。
a.基于 [U-¹³C]-葡萄糖 标记不同时间后单细胞 LE 特征的 UMAP 分析。Z-score 归一化,总细胞数 n = 5,160。
b.对 (a) 中全部细胞进行DBSCAN 聚类,得到 A–F 共 6 个亚群(eps = 1,min_samples = 100)。
c.6 个标记时间点单细胞在 A–F 亚群中的分布。每图下方标注该时间点细胞数。0 min 图中虚线框内容在其他子图中放大显示。
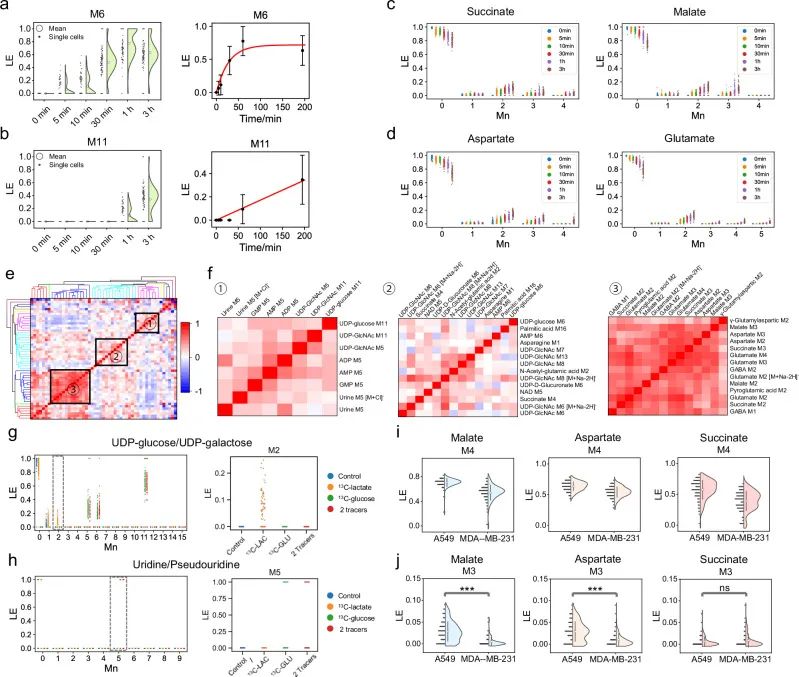
图5. 在单细胞水平上,通过多功能标记示踪剂实现代谢流动和相互连接。
a-b. 分别展示 (a) M6 和 (b) M11 标记的 UDP-葡萄糖/UDP-半乳糖随时间变化标记模式。左:单细胞小提琴图;右:均值±SD(黑点)及指数拟合红线。每组 n = 100 细胞。
c-d. 分别展示 (c) TCA 循环中间产物与 (d) 相关代谢物的时序标记模式。每组 n = 100 细胞。
e.3 h 标记后单细胞 LE 的 Pearson 相关系数矩阵(剔除含零值细胞)。
f.(e) 中相关系数矩阵的局部放大图,n = 268。
g-h. 分别用 [U-¹³C]-乳酸、[U-¹³C]-葡萄糖及双示踪剂标记 12 h 后 (g) UDP-葡萄糖/UDP-半乳糖 和 (h) 尿苷/假尿苷 的标记模式。蓝、橙、绿、红点分别对应无标记、[U-¹³C]-乳酸、[U-¹³C]-葡萄糖、双示踪剂组,每组 n = 200 细胞。
i-j. 分别用 [U-¹³C]-谷氨酰胺标记 3 h 后 (i) M4 和 (j) M3 标记代谢物在 A549 与 MDA-MB-231 细胞中的 LE。数据为均值±SD,黑点为单细胞值;双侧 Wilcoxon 秩和检验,***P < 0.001,ns 为无显著差异。每组 n = 100 细胞。
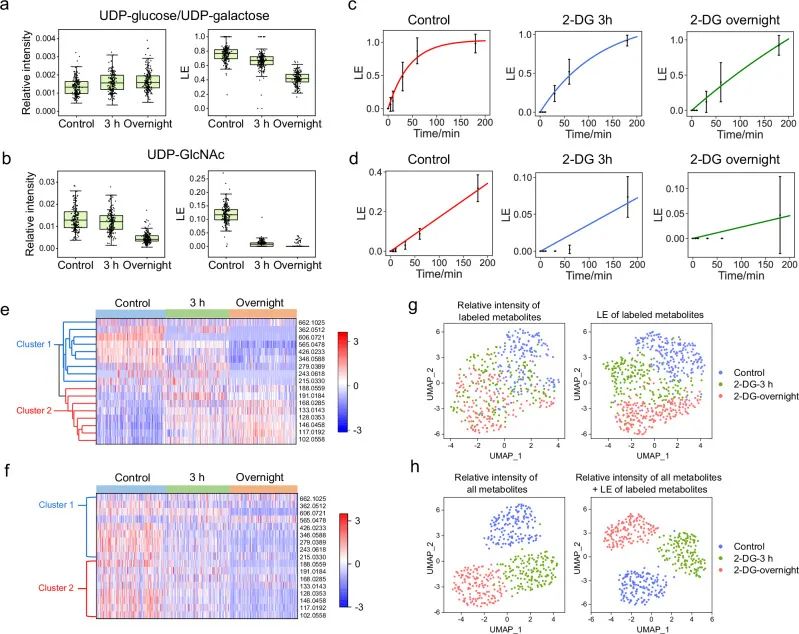
图6. 2-DG诱导的代谢活性扰动与单细胞聚类分析。
a.三组细胞中 UDP-葡萄糖/UDP-半乳糖的相对强度与 LE 比较。
b.三组细胞中 UDP-GlcNAc 的相对强度与 LE 比较。箱线定义同前,每组 n = 200 细胞。
c-d. 分别展示 (c) UDP-葡萄糖/UDP-半乳糖 和 (d) UDP-GlcNAc 在三组中的 3 h 时序标记曲线。数据为均值±SD,曲线为指数拟合,每组 n = 100 细胞。
e.标记代谢物 LE(Z-score 归一化)的分层聚类。
f.标记代谢物相对强度(Z-score 归一化)热图,每组 n = 200 细胞。
g.基于相对强度或标记代谢物 LE 的三组单细胞 UMAP 分析,细胞数分别为 150 与 200。
h.基于全部代谢物相对强度 ± 标记代谢物 LE 的三组 UMAP 分析,每组 n = 150 细胞。
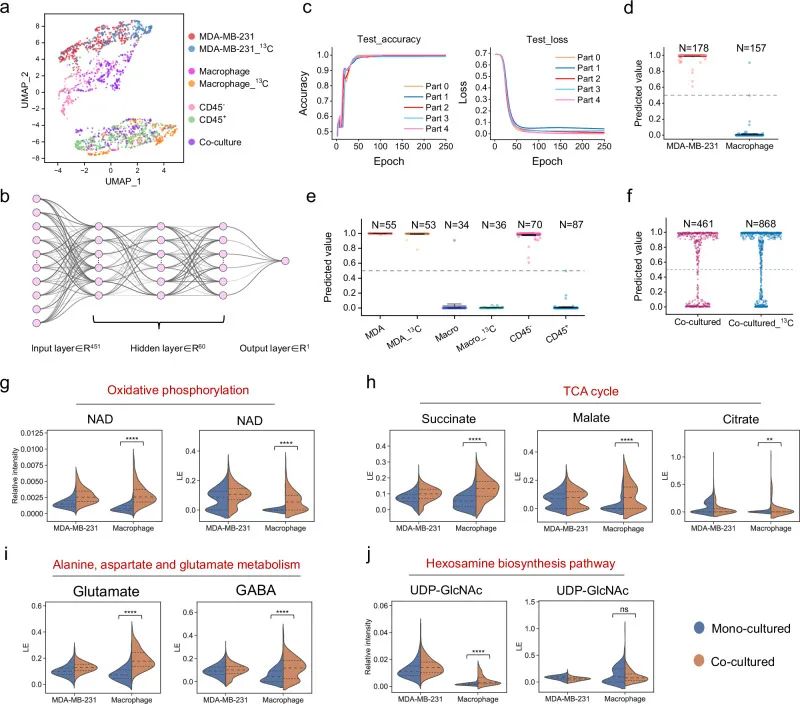
图7. 共培养MDA-MB-231细胞和巨噬细胞的代谢组学分析。
a.单细胞 UMAP 分析。细胞数:MDA-MB-231 = 334,巨噬细胞 = 65,¹³C 标记 MDA-MB-231 = 282,¹³C 标记巨噬细胞 = 204,CD45⁻ = 297,CD45⁺ = 470,共培养 = 327。FACS 分选门控见Source Data。
b.神经网络模型结构示意图。
c.测试数据集准确率与损失函数随训练周期变化曲线。
d-e. 分别展示 (d) 整个测试集与 (e) 其各子集的预测结果。
f.共培养样本的预测结果。
g–j. 共培养与单培养细胞浓度及活性变化比较。小提琴图中虚线为中位数,上下为 25%/75% 分位数。双侧 Wilcoxon 秩和检验,显著性 P 值已在图中标注。|Fold Change| > 2 的组标出 P 值。细胞数:单培养 MDA-MB-231 = 794,¹³C 标记单培养 MDA-MB-231 = 833,单培养巨噬细胞 = 245,¹³C 标记单培养巨噬细胞 = 223,共培养 MDA-MB-231 = 303,共培养巨噬细胞 = 158,¹³C 标记共培养 MDA-MB-231 = 635,¹³C 标记共培养巨噬细胞 = 233。
参考文献:
Zhang Y, Shi M, Li M, Qin S, Miao D, Bai Y. Dynamic single-cell metabolomics reveals cell-cell interaction between tumor cells and macrophages. Nat Commun. 2025 May 16;16(1):4582. doi: 10.1038/s41467-025-59878-w. PMID: 40379657; PMCID: PMC12084531.